CRISPR y coronavirus
Por Lluis Montoliu, el 3 abril, 2020. Categoría(s): diagnóstico genético • edición genética • genética ✎ 41
Los que me hayáis escuchado alguna vez impartiendo una charla sobre CRISPR, o hayáis leído mi libro «Editando genes: recorta, pega y colorea. Las maravillosas herramientas CRISPR» (NextDoor Publishers, 2019) ya sabréis que una de las frases que más repito yo es: «la imaginación es el límite de las aplicaciones CRISPR«. Por eso no nos debería sorprender que el universo CRISPR también tenga algo que decir en la crisis actual sanitaria mundial causada por el coronavirus SARS-CoV-2, causante de la COVID-19.
En este artículo explicaré los dos grandes grupos de aplicaciones CRISPR para DIAGNOSTICAR y para COMBATIR el coronavirus.
Diagnóstico del coronavirus mediante CRISPR
La primera «aplicación» CRISPR para el nuevo coronavirus vino de la mano del laboratorio de Feng Zhang (BROAD-MIT, Boston, MS, USA), el inventor de la técnica de diagnóstico mediante CRISPR llamada SHERLOCK (nombre con evidente gancho y doble sentido, que es un acrónimo de las palabras en inglés Specific High-sensitivity Enzymatic Reporter unLOCKing), y que su laboratorio describió en 2017. El diagnóstico mediante CRISPR y la técnica SHERLOCK está ilustrado en la figura que encabeza este artículo. Esencialmente el protocolo se basa en una nueva proteína efectora Cas, que ya no es Cas9 sino que es Cas13a, de otra bacteria. La Cas13a tiene la capacidad de cortar ARN (y no ADN, como Cas9), y de activarse gracias a una pequeña guía de ARN específica que sea complementaria al ARN que se quiere cortar y degradar. El grupo de Feng Zhang encontró que al activarse in vitro (en el laboratorio) esta RNAsa (nucleasa que degrada el ARN) esta parecía volverse loca y acababa cortando y degradando no solamente el ARN complementario diana sino todos los ARN que hubiera en el ensayo. Este hallazgo, que hubiera sido interpretado como un gran fiasco por la mayoría de investigadores, despertó la perspicacia y el talento de Feng Zhang, y acabó convirtiendo un resultado negativo inesperado en una nueva aplicación para diagnosticar la presencia de moléculas de ADN (y ARN) presentes en ínfimas cantidades en una muestra.
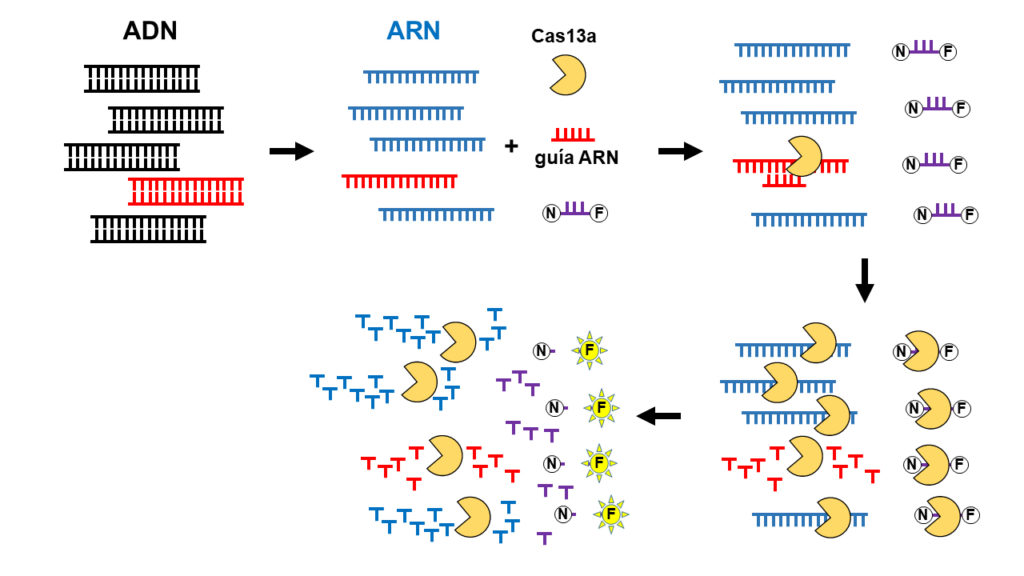
La idea magistral que se oculta tras SHERLOCLK es la adición de unas pequeñas moléculas de ARN (de color morado en la ilustración de cabecera) que tienen en uno de sus extremos una moléculas fluorescente (F) y en el otro extremo una molécula inhibidora de esa fluorescencia (N). Cuando las dos moléculas F y N están juntas no se emite fluorescencia. Tras activarse el corte del ARN diana por parte de la proteína Cas13a, gracias a una guía específica de ARN, complementaria al ARN a detectar, la Cas13a no solo corta ese ARN sino todos los presentes en la mezcla, incluyendo las pequeñas moléculas de ARN de color morado. Estas, al partirse, liberarán las moléculas F y N por separado y, entonces, la molécula F podrá brillar y mostrar su fluorescencia, siendo posible detectar este brillo de luz mediante detectores lumínicos específicos. Dado que la fluorescencia no aparece hasta que se inicie la degradación de los ARN y, dado que esta degradación no se inicia si no es en presencia del ARN complementario a la guía ARN específica, el sistema SHERLOCK representa un método muy específico y sensible (se estima su sensibilidad en el orden de attomolar, esto es detecta una molécula de ARN que esté diluida hasta una concentración de 10[exp -18] molar) para detectar un ADN (que debe ser convertido primero a ARN mediante una transcripción in vitro) o un ARN (que no necesita ese primer paso y puede aplicarse directamente).
Efectivamente, a principios de este año, y tras conocerse la secuencia del genoma ARN del coronavirus SARS-CoV-2, causante de la COVID-19, el laboratorio de Feng Zhang hizo público un protocolo de detección del coronavirus mediante SHERLOCK y compartió el protocolo y los detalles técnicos para llevarlo a cabo en una publicación abierta a todo el mundo. El protocolo es relativamente sencillo (si se tienen todos los reactivos, fácilmente obtenibles desde Addgene y otros proveedores) y puede completarse en apenas 1 hora. La sensibilidad del método permite detectar hasta 10-100 moléculas del genoma del coronavirus por microlitro (~20-200 aM) Como ellos mismos indican en su protocolo de detección, este no está todavía homologado ni autorizado para aplicarlo en el diagnóstico clínico del coronavirus (algo que tendrá que aprobar eventualmente la FDA, tras realizar las revisiones y análisis correspondientes), pero si puede usarse de forma experimental, en los laboratorios. La empresa creada por Zhang «Sherlock Biosciences» está desarrollando el kit de detección y esperando poder aplicar esta tecnología para realizar diagnósticos masivos del coronavirus SARS-CoV-2 mediante SHERLOCK.
Párrafo añadido el 5 de mayo de 2020: Feng Zhang acaba de hacer público un nuevo método de diagnóstico optimizado y simplificado, derivado de SHERLOCK, aplicable para detectar el coronavirus SARS-CoV-2 directamente, sin mediar extracción o aislamiento de ARN, que apenas requiere de 40-70 minutos para obtenerse el resultado, tras una incubación a 60 grados. El nuevo método ha recibido el nombre de STOPCovid y puede ser un nuevo revulsivo en el paisaje actual de técnicas diagnósticas rápidas para detectar el genoma del virus de formas mucho más rápidas y simples que mediante RT-PCR. El límite de detección de este método es de unas 100 copias del ARN del coronavirus por reacción. En STOPCovid los autores combinan la técnica LAMP (siglas de Amplificación Isotérmica mediada por lazos) con el uso de la variante CRISPR-Cas12b
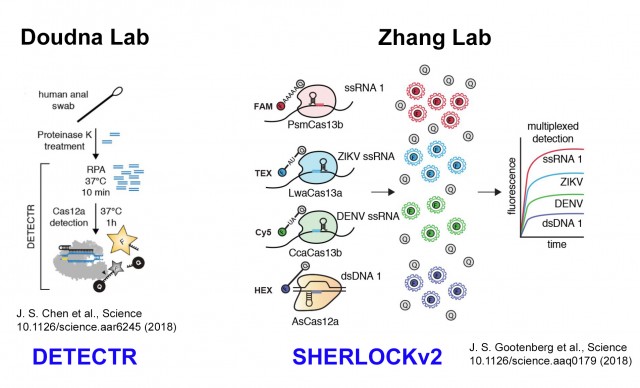
A principios de 2018, el laboratorio de Jennifer Doudna (UC Berkeley, CA, USA), una de las pioneras de la revolución CRISPR y sus aplicaciones en edición genética, desarrolló un test de diagnóstico genético CRISPR análogo a SHERLOCK pero basado en otra proteína Cas con propiedades similares a Cas13a. En este caso se trataba de la proteína Cas12a y al método resultante lo bautizaron como DETECTR (nombre también con doble sentido y cuidadosamente elegido, que es un acrónimo de las palabras en inglés DNA Endonuclease TargEted CRISPR Trans Reporter). En paralelo, el laboratorio de Feng Zhang respondió combinando hasta cuatro proteínas Cas con actividades RNAsa (Cas13b de dos bacterias distintas, Cas13a y Cas12a) para detectar hasta cuatro moléculas de ADN (o ARN) distintas, en un desarrollo tecnológico que llamó naturalmente como SHERLOCKv2. En una entrada anterior de este blog comparé y expliqué en detalle los dos sistemas de diagnóstico basados en CRISPR: DETECTR y SHERLOCK.
Naturalmente el sistema DETECTR también puede aplicarse para detectar el coronavirus SARS-CoV-2. Han aparecido publicaciones que usan la proteína Cas12a para diagnosticar la presencia del coronavirus de forma rápida, sencilla y asequible. Alguno de estos métodos que usan la proteína Cas12a es capaz de detectar el virus HIV, causante del SIDA, y el coronavirus SARS-CoV-2, simultáneamente. La empresa fundada por Jennifer Doudna, Mammoth Biosciences, de igual forma que Sherlock Biosciences, también está desarrollando sistemas de detección del coronavirus basados en la tecnología DETECTR, que anuncian que pueden ser todavía más rápidos, simples y programables.
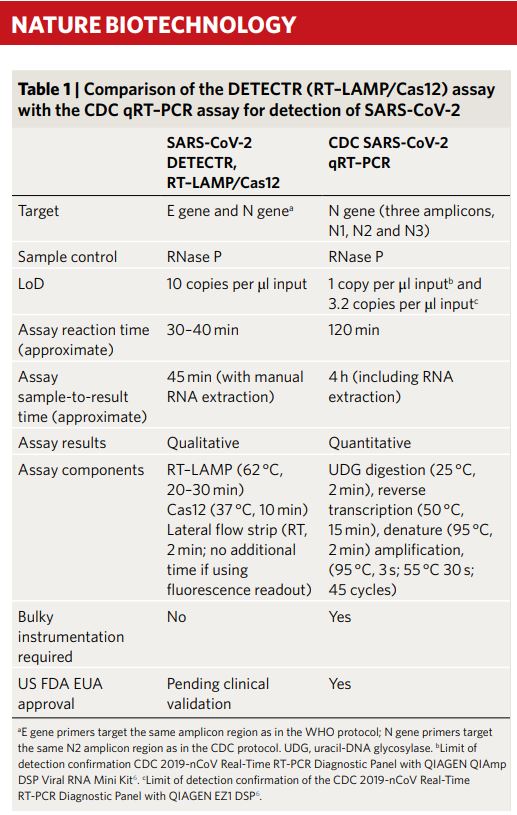
Párrafo y tabla adjunta añadidos el 17 de abril de 2020: Ayer se publicó en la revista Nature Biotechnology el método que propone la empresa Mammoth Biosciences, basado en DETECTR, para detectar la presencia del virus SARS-CoV-2 mediante Cas12a. La tabla comparativa adjunta, que se incluye en este artículo, muestra las ventajas e inconvenientes de DETECR frente a la RT-PCR. En resumen, es más rápido (45 minutos por unas 3-4 horas) pero es menos sensible (10 copias del virus por microlitro frente a 1 copia del genoma viral que es capaz de detectar la RT-PCR). La diferencia adicional es que SHERLOCK puede procesar directamente la muestra del coronavirus (dado que Cas13a corta ARN) mientras que DETECTR necesita una etapa previa de retrotranscripción, de conversión de ARN a ADN, puesto que Cas12a corta ADN, no ARN. En ambos casos hay una fase de amplificación previa antes de cortar la secuencia problema.
De cualquier manera, estos métodos innovadores de diagnóstico de la presencia del genoma ARN del coronavirus mediante SHERLOCK o DETECTR respresentan la punta de lanza biotecnológica de los diagnósticos genéticos basados en CRISPR, y seguramente serán la respuesta que necesitamos para poder detectar este virus de forma masiva, rápida y sencilla. Por el momento, en un listado en el que aparecen la mayoría de test de diagnóstico de SARS-CoV-2 comerciales o en desarrollo solo se menciona un kit de diagnóstico, en desarrollo, basado en la tecnología CRISPR.
Combatir al coronavirus mediante CRISPR
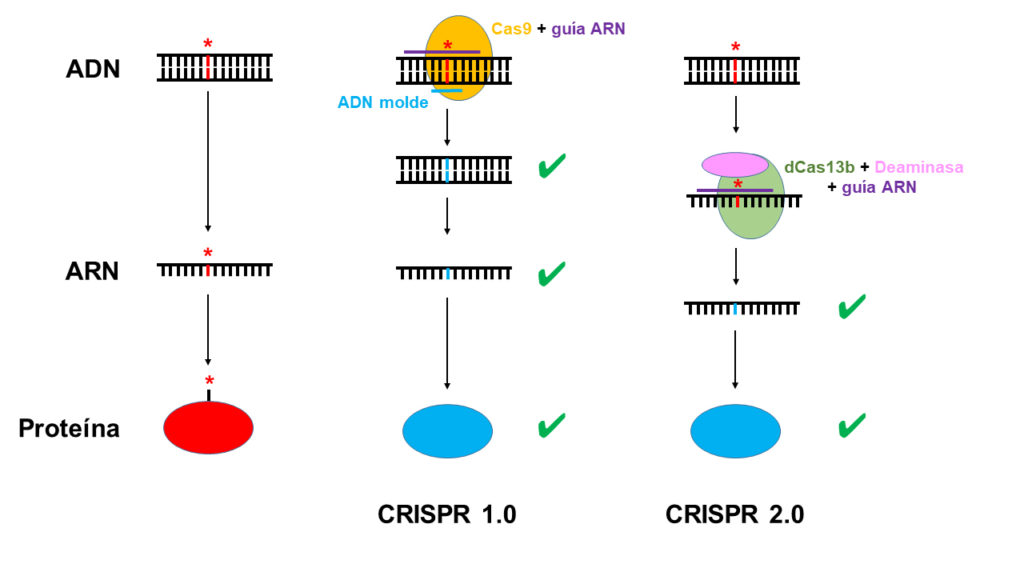
Generalmente todos tenemos en mente que las herramientas CRISPR son capaces de editar cualquier secuencia de ADN. Y esto es cierto. Las variantes más comunmente utilizadas (CRISPR-Cas9) cortan y promueven la edición de ADN. Son las llamadas herramientas CRISPR 1.0. Adicionalmente, como he explicado en el apartado anterior, existen diversas proteínas Cas con capacidad de cortar ARN de forma específica. Algunas de ellas, como la Cas13b, han sido modificadas en el laboratorio, eliminando su capacidad de corte y combinándolas con actividades desaminasas que son capaces de cambiar directamente, químicamente, algún ribonucleótido específico (alguna letra concreta del ARN, la A por la G, o la C por la U), permitiendo la edición directa del ARN, lo que se ha venido a llamar las herramientas de edición CRISPR 2.0, como están ilustradas en la figura anterior. Estas herramientas (de las que existen diversas modalidades ya disponibles) podrían usarse, por ejemplo, para alterar la secuencia del genoma ARN del coronavirus SARS-CoV-2 mutando aquellos genes que le confieren la virulencia y, por ello, convirtiéndose en herramientas CRISPR para combatir la infección del coronavirus.
Sin embargo, una forma todavía más efectiva de combatir al coronavirus SARS-CoV-2 mediante CRISPR es precisamente usando la capacidad que tienen algunas de estas proteínas Cas13 para cortar, degradar y destruir moléculas de ARN específicas. En particular usando la proteína Cas13d, con actividad RNAsa específica, guiada por una pequeña molécula de RNA.
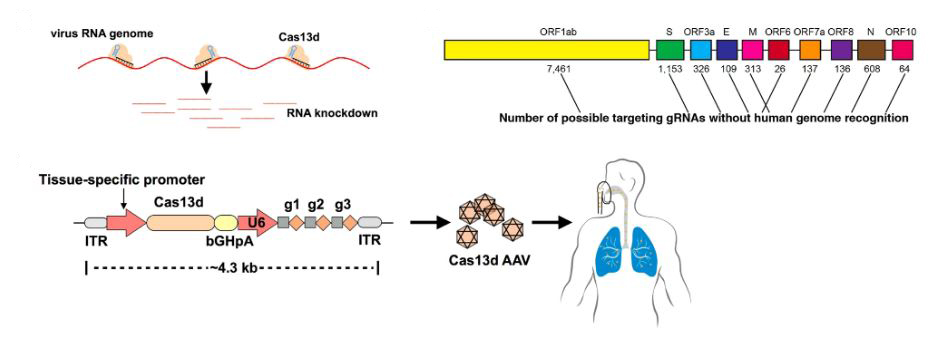
En febrero de este año apareció publicado en la revista Cell Research un artículo, lanzado desde la Facultad de Medicina de Harvard, en EE.UU., que postulaba un diseño terapéutico basado en Cas13d para combatir el coronavirus mediante CRISPR. Los autores de la propuesta habían localizado múltiples dianas para usar como complementarias a las guías ARN de la proteína Cas13d en el genoma del coronavirus SARS-CoV-2. Su propuesta incluía introducir dentro de partículas virales AAV (virus adeno-asociados, muy utilizados en terapia génica) el gen que codifica la proteína Cas13d y un bloque de expresión de tres guías de ARN que usaría la Cas13d para cortar el genoma del coronavirus. El AAV recombinante resultante podría administrarse por vía aérea, para que llegará fácilmente a los pulmones, entrara en las células cargadas de coronavirus y los destruyera. En teoría. Ahora falta demostrar en la práctica toda esta propuesta, de momento lanzada como una propuesta interesante, innovadora, pero teórica, todavía en desarrollo.
Otro estudio, este sí ya completado experimentalmente, desarrollado en la costa oeste de EE.UU., en la Universidad de Stanford, acaba de ser depositado en el servidor de pre-prints bioRxiv, el pasado 14 de marzo de 2020, y en él los investigadores también usan la estrategia de Cas13d para combatir a diferentes virus RNA. En este trabajo los autores prepararon una línea de células humanas epiteliales de pulmón a las que previamente transformaron con una construcción génica para que produjeran constantemente proteína Cas13d y un marcador fluorescente. Posteriormente estas células las transfectaban con nuevas construcciones capaces de producir las guías RNA que necesita la proteína Cas13d para cortar el ARN en posiciones específicas y exponían las células o bien a construcciones que simulan la infección con el coronavirus SARS-CoV-2 o a virus de la gripe (IAV), que también tiene moléculas de ARN en su genoma. En ambos casos tuvieron éxito, consiguiendo una degradación de las secuencias del ARN del SARS-CoV-2 y una inhibición en la replicación del virus de la gripe. Los autores bautizaron su método con el no menos ingenioso nombre de PAC-MAN (acrónimo de las palabras en inglés Prophylactic Antiviral CRISPR in huMAN cells). Los autores realizaron un análisis comparativo de los genomas de todos los coronavirus humanos conocidos y llegaron a encontrar seis moléculas de ARN que pueden actuar como guía para la proteína Cas13d, capaces de aparearse con secuencias comunes a todos los coronavirus conocidos que nos infectan a los seres humanos, lo cual, de poder verificarse, representaría una arma antiviral profiláctica y terapéutica extraordinariamente vesátil y poderosa, basada en herramientas CRISPR.
Aunque sabemos poco de las nuevas versiones CRISPR, de estas proteínas Cas13d, ya sabemos que pueden funcionar también bien in vivo, en animales. Miguel Ángel Moreno Mateos, investigador Ramón y Cajal de la Universidad Pablo de Olavide, en el Centro Andaluz de Biología del Desarrollo (CABD) en Sevilla, ha demostrado, en un estudio reciente, depositado en bioRxiv el 14 de enero de 2020, que este sistema CRISPR-Cas13d funciona para reducir la expresión de genes específicos en embriones de pez cebra, de pez medaka, de pez killi y de ratón. Unos datos muy interesantes que permiten albergar esperanzas para su futuro uso en animales adultos y, eventualmente, si todos los análisis previos fueran exitosos, en personas.
Las herramientas CRISPR no dejan de sorprendernos. Han aparecido en los laboratorios, apenas hace siete años, y han venido para quedarse. Y la imaginación desbordante y sin límites de los investigadores hace el resto. ¡Larga vida a las CRISPR! ¡Mucho éxito diagnosticando y combatiendo a los coronavirus!
Este artículo ha sido actualizado dos veces: el 17 de abril de 2020 y el 5 de mayo de 2020.


